Pandas - трюки и возможности, о которых вы, возможно, не знаете
Оглавление
- 1. Настройка опций и параметров при запуске интерпретатора
- 2. Создавайте игрушечные структуры данных с помощью модуля тестирования pandas
- 3. Используйте преимущества методов-акселераторов
- 4. Создание индекса времени даты из столбцов компонентов
- 5. Использование категориальных данных для экономии времени и места
- 6. Интроспекция объектов Groupby с помощью итерации
- 7. Используйте этот трюк с отображением для биннинга членства
- 8. Поймите, как pandas использует булевы операторы
- 9. Загрузка данных из буфера обмена
- 10. Запись объектов pandas непосредственно в сжатый формат
Pandas - это фундаментальная библиотека для аналитики, обработки данных и науки о данных. Это огромный проект с огромным количеством возможностей и глубиной.
В этом уроке мы рассмотрим некоторые менее используемые, но идиоматические возможности pandas, которые повышают читабельность, универсальность и скорость вашего кода, а-ля листик из Buzzfeed.
Если вы хорошо знакомы с основными концепциями библиотеки Python pandas, надеемся, что в этой статье вы найдете пару трюков, на которые не натыкались ранее. (Если вы только начинаете знакомиться с библиотекой, то для начала подойдет книга 10 Minutes to pandas)
1. Настройка параметров и настроек при запуске переводчика
Возможно, вы уже сталкивались с богатой системой опций и настроек Pandas.
Огромную экономию производительности дает установка настраиваемых параметров pandas при запуске интерпретатора, особенно если вы работаете в среде сценариев. Вы можете использовать pd.set_option() для настройки по своему усмотрению с помощью Python или IPython стартового файла.
В опциях используется точечная нотация, например pd.set_option('display.max_colwidth', 25), которая хорошо подходит для вложенного словаря опций:
import pandas as pd
def start():
options = {
'display': {
'max_columns': None,
'max_colwidth': 25,
'expand_frame_repr': False, # Don't wrap to multiple pages
'max_rows': 14,
'max_seq_items': 50, # Max length of printed sequence
'precision': 4,
'show_dimensions': False
},
'mode': {
'chained_assignment': None # Controls SettingWithCopyWarning
}
}
for category, option in options.items():
for op, value in option.items():
pd.set_option(f'{category}.{op}', value) # Python 3.6+
if __name__ == '__main__':
start()
del start # Clean up namespace in the interpreter
Если вы запустите сеанс интерпретатора, то увидите, что все, что написано в стартовом скрипте, уже выполнено, а pandas импортируется автоматически с вашим набором опций:
>>> pd.__name__
'pandas'
>>> pd.get_option('display.max_rows')
14
Для демонстрации форматирования, заданного в файле запуска, воспользуемся некоторыми данными по abalone, размещенными в UCI Machine Learning Repository. Данные будут усечены до 14 строк с точностью 4 знака для плавающих чисел:
>>> url = ('https://archive.ics.uci.edu/ml/'
... 'machine-learning-databases/abalone/abalone.data')
>>> cols = ['sex', 'length', 'diam', 'height', 'weight', 'rings']
>>> abalone = pd.read_csv(url, usecols=[0, 1, 2, 3, 4, 8], names=cols)
>>> abalone
sex length diam height weight rings
0 M 0.455 0.365 0.095 0.5140 15
1 M 0.350 0.265 0.090 0.2255 7
2 F 0.530 0.420 0.135 0.6770 9
3 M 0.440 0.365 0.125 0.5160 10
4 I 0.330 0.255 0.080 0.2050 7
5 I 0.425 0.300 0.095 0.3515 8
6 F 0.530 0.415 0.150 0.7775 20
# ...
4170 M 0.550 0.430 0.130 0.8395 10
4171 M 0.560 0.430 0.155 0.8675 8
4172 F 0.565 0.450 0.165 0.8870 11
4173 M 0.590 0.440 0.135 0.9660 10
4174 M 0.600 0.475 0.205 1.1760 9
4175 F 0.625 0.485 0.150 1.0945 10
4176 M 0.710 0.555 0.195 1.9485 12
Позже вы увидите этот набор данных и в других примерах.
2. Создание игрушечных структур данных с помощью модуля тестирования pandas
Примечание: Модуль pandas.util.testing был устаревшим в pandas 1.0. Теперь "публичный API для тестирования" из pandas.testing ограничен assert_extension_array_equal(), assert_frame_equal(), assert_series_equal() и assert_index_equal(). Автор признает, что получает по заслугам за то, что полагается на недокументированные части библиотеки pandas.
Внизу в модуле pandas testing находится ряд удобных функций для быстрого построения квазиреалистичных серий и фреймов данных:
>>> import pandas.util.testing as tm
>>> tm.N, tm.K = 15, 3 # Module-level default rows/columns
>>> import numpy as np
>>> np.random.seed(444)
>>> tm.makeTimeDataFrame(freq='M').head()
A B C
2000-01-31 0.3574 -0.8804 0.2669
2000-02-29 0.3775 0.1526 -0.4803
2000-03-31 1.3823 0.2503 0.3008
2000-04-30 1.1755 0.0785 -0.1791
2000-05-31 -0.9393 -0.9039 1.1837
>>> tm.makeDataFrame().head()
A B C
nTLGGTiRHF -0.6228 0.6459 0.1251
WPBRn9jtsR -0.3187 -0.8091 1.1501
7B3wWfvuDA -1.9872 -1.0795 0.2987
yJ0BTjehH1 0.8802 0.7403 -1.2154
0luaYUYvy1 -0.9320 1.2912 -0.2907
Их около 30, и вы можете увидеть полный список, вызвав dir() на объекте модуля. Вот некоторые из них:
>>> [i for i in dir(tm) if i.startswith('make')]
['makeBoolIndex',
'makeCategoricalIndex',
'makeCustomDataframe',
'makeCustomIndex',
# ...,
'makeTimeSeries',
'makeTimedeltaIndex',
'makeUIntIndex',
'makeUnicodeIndex']
Это может быть полезно для бенчмаркинга, тестирования утверждений и экспериментов с методами pandas, с которыми вы менее знакомы.
3. Используйте преимущества методов доступа
Возможно, вы слышали о термине accessor, который чем-то напоминает геттер (хотя геттеры и сеттеры в Python используются нечасто). Для наших целей здесь можно представить себе аксессор pandas как свойство, которое служит интерфейсом для дополнительных методов.
>>> pd.Series._accessors
{'cat', 'str', 'dt'}
Да, приведенное выше определение многозначно, поэтому давайте рассмотрим несколько примеров, прежде чем обсуждать внутреннее устройство.
.cat предназначен для категориальных данных, .str - для строковых (объектных) данных, а .dt - для данных, похожих на время. Начнем с .str: представьте, что у вас есть необработанные данные о городе/штате/IP как одно поле в pandas Series.
Строковые методы pandas являются векторизованными, что означает, что они работают со всем массивом без явного for цикла:
>>> addr = pd.Series([
... 'Washington, D.C. 20003',
... 'Brooklyn, NY 11211-1755',
... 'Omaha, NE 68154',
... 'Pittsburgh, PA 15211'
... ])
>>> addr.str.upper()
0 WASHINGTON, D.C. 20003
1 BROOKLYN, NY 11211-1755
2 OMAHA, NE 68154
3 PITTSBURGH, PA 15211
dtype: object
>>> addr.str.count(r'\d') # 5 or 9-digit zip?
0 5
1 9
2 5
3 5
dtype: int64
Для более сложного примера предположим, что вы хотите разделить три компонента city/state/ZIP аккуратно на поля DataFrame.
Вы можете передать регулярное выражение в .str.extract(), чтобы "извлечь" части каждой ячейки в Серии. В .str.extract(), .str - это аксессор, а .str.extract() - метод аксессора:
>>> regex = (r'(?P<city>[A-Za-z ]+), ' # One or more letters
... r'(?P<state>[A-Z]{2}) ' # 2 capital letters
... r'(?P<zip>\d{5}(?:-\d{4})?)') # Optional 4-digit extension
...
>>> addr.str.replace('.', '').str.extract(regex)
city state zip
0 Washington DC 20003
1 Brooklyn NY 11211-1755
2 Omaha NE 68154
3 Pittsburgh PA 15211
Это также иллюстрирует то, что известно как цепочка методов, где .str.extract(regex) вызывается на результат addr.str.replace('.', ''), который очищает использование периодов, чтобы получить красивую двухсимвольную аббревиатуру государства.
Полезно знать немного о том, как работают эти методы-аксессоры, чтобы мотивировать, почему вы должны использовать их в первую очередь, а не что-то вроде addr.apply(re.findall, ...).
Каждый аксессуар сам по себе является добросовестным классом в Python:
.strотображается наStringMethods..dtкарты наCombinedDatetimelikeProperties..catмаршруты вCategoricalAccessor.
Затем эти автономные классы "прикрепляются" к классу Series с помощью CachedAccessor. Именно тогда, когда классы обернуты в CachedAccessor, происходит немного волшебства.
CachedAccessor вдохновлен дизайном "кэшированного свойства": свойство вычисляется только один раз для экземпляра, а затем заменяется обычным атрибутом. Для этого используется перегрузка метода .__get__() , который является частью протокола Python descriptor.
Примечание: Если вы хотите прочитать больше о том, как это работает, смотрите Python Descriptor HOWTO и этот пост о дизайне кэшированных свойств. В Python 3 также появилась функция functools.lru_cache(), которая предлагает аналогичную функциональность. Примеры этого паттерна можно найти повсюду, например, в пакете aiohttp.
Второй аксессор, .dt, предназначен для данных, похожих на время. Технически он принадлежит к DatetimeIndex Панды, и если его вызвать для серии, он сначала преобразуется в DatetimeIndex:
>>> daterng = pd.Series(pd.date_range('2017', periods=9, freq='Q'))
>>> daterng
0 2017-03-31
1 2017-06-30
2 2017-09-30
3 2017-12-31
4 2018-03-31
5 2018-06-30
6 2018-09-30
7 2018-12-31
8 2019-03-31
dtype: datetime64[ns]
>>> daterng.dt.day_name()
0 Friday
1 Friday
2 Saturday
3 Sunday
4 Saturday
5 Saturday
6 Sunday
7 Monday
8 Sunday
dtype: object
>>> # Second-half of year only
>>> daterng[daterng.dt.quarter > 2]
2 2017-09-30
3 2017-12-31
6 2018-09-30
7 2018-12-31
dtype: datetime64[ns]
>>> daterng[daterng.dt.is_year_end]
3 2017-12-31
7 2018-12-31
dtype: datetime64[ns]
Третий аксессуар, .cat, предназначен только для категориальных данных, которые вы вскоре увидите в его собственном разделе.
4. Создание индекса времени даты из столбцов компонентов
Говоря о данных, похожих на время, как в daterng выше, можно создать pandas DatetimeIndex из нескольких столбцов-компонентов, которые вместе образуют дату или время данных:
>>> from itertools import product
>>> datecols = ['year', 'month', 'day']
>>> df = pd.DataFrame(list(product([2017, 2016], [1, 2], [1, 2, 3])),
... columns=datecols)
>>> df['data'] = np.random.randn(len(df))
>>> df
year month day data
0 2017 1 1 -0.0767
1 2017 1 2 -1.2798
2 2017 1 3 0.4032
3 2017 2 1 1.2377
4 2017 2 2 -0.2060
5 2017 2 3 0.6187
6 2016 1 1 2.3786
7 2016 1 2 -0.4730
8 2016 1 3 -2.1505
9 2016 2 1 -0.6340
10 2016 2 2 0.7964
11 2016 2 3 0.0005
>>> df.index = pd.to_datetime(df[datecols])
>>> df.head()
year month day data
2017-01-01 2017 1 1 -0.0767
2017-01-02 2017 1 2 -1.2798
2017-01-03 2017 1 3 0.4032
2017-02-01 2017 2 1 1.2377
2017-02-02 2017 2 2 -0.2060
Наконец, вы можете отказаться от старых отдельных столбцов и преобразовать их в серию:
>>> df = df.drop(datecols, axis=1).squeeze()
>>> df.head()
2017-01-01 -0.0767
2017-01-02 -1.2798
2017-01-03 0.4032
2017-02-01 1.2377
2017-02-02 -0.2060
Name: data, dtype: float64
>>> df.index.dtype_str
'datetime64[ns]
Интуиция передачи DataFrame заключается в том, что DataFrame похож на словарь Python, где имена столбцов являются ключами, а отдельные столбцы (Series) - значениями словаря. Поэтому pd.to_datetime(df[datecols].to_dict(orient='list')) также будет работать в этом случае. Это повторяет конструкцию питоновского datetime.datetime, где вы передаете аргументы с ключевыми словами, такими как datetime.datetime(year=2000, month=1, day=15, hour=10).
5. Использование категориальных данных для экономии времени и места
Одной из мощных возможностей pandas является ее Categorical dtype.
Даже если вы не всегда работаете с гигабайтами данных в оперативной памяти, вы наверняка сталкивались со случаями, когда простые операции над большим DataFrame зависали более чем на несколько секунд.
pandas object dtype часто является отличным кандидатом для преобразования в категорию данных. (object - это контейнер для Python str, разнородных типов данных или "других" типов). Строки занимают значительное место в памяти:
>>> colors = pd.Series([
... 'periwinkle',
... 'mint green',
... 'burnt orange',
... 'periwinkle',
... 'burnt orange',
... 'rose',
... 'rose',
... 'mint green',
... 'rose',
... 'navy'
... ])
...
>>> import sys
>>> colors.apply(sys.getsizeof)
0 59
1 59
2 61
3 59
4 61
5 53
6 53
7 59
8 53
9 53
dtype: int64
Примечание: я использовал sys.getsizeof(), чтобы показать память, занимаемую каждым отдельным значением в серии. Не забывайте, что это объекты Python, которые имеют некоторые накладные расходы. (sys.getsizeof('') вернет 49 байт.)
Существует также colors.memory_usage(), который суммирует использование памяти и полагается на атрибут .nbytes базового массива NumPy. Не стоит слишком увлекаться этими деталями: важно относительное использование памяти, которое возникает в результате преобразования типов, как вы увидите далее.
Теперь, что если мы могли бы взять уникальные цвета, перечисленные выше, и отобразить каждый из них в целое число, занимающее меньше места? Вот наивная реализация этого:
>>> mapper = {v: k for k, v in enumerate(colors.unique())}
>>> mapper
{'periwinkle': 0, 'mint green': 1, 'burnt orange': 2, 'rose': 3, 'navy': 4}
>>> as_int = colors.map(mapper)
>>> as_int
0 0
1 1
2 2
3 0
4 2
5 3
6 3
7 1
8 3
9 4
dtype: int64
>>> as_int.apply(sys.getsizeof)
0 24
1 28
2 28
3 24
4 28
5 28
6 28
7 28
8 28
9 28
dtype: int64
Примечание: Другой способ сделать то же самое - использовать pd.factorize(colors) панды:
>>> pd.factorize(colors)[0]
array([0, 1, 2, 0, 2, 3, 3, 1, 3, 4])
В любом случае вы кодируете объект как перечислимый тип (категориальная переменная).
Вы сразу заметите, что использование памяти сократилось почти вдвое по сравнению с использованием полных строк с помощью object dtype.
Ранее в разделе о акцессорах я упоминал .cat (категориальный) акцессор. Вышеприведенное с mapper является грубой иллюстрацией того, что происходит внутри пандасовского Categorical dtype:
"Использование памяти
Categoricalпропорционально количеству категорий плюс длина данных. В отличие от этого, dtypeobjectпредставляет собой константу, умноженную на длину данных." (Источник)
В colors выше, у вас есть соотношение 2 значений для каждого уникального значения (категории):
>>> len(colors) / colors.nunique()
2.0
В результате экономия памяти при преобразовании в Categorical хороша, но не велика:
>>> # Not a huge space-saver to encode as Categorical
>>> colors.memory_usage(index=False, deep=True)
650
>>> colors.astype('category').memory_usage(index=False, deep=True)
495
Однако если раздуть вышеупомянутую пропорцию при большом количестве данных и малом количестве уникальных значений (вспомните данные по демографии или алфавитные результаты тестов), то требуемое сокращение памяти будет более чем в 10 раз:
>>> manycolors = colors.repeat(10)
>>> len(manycolors) / manycolors.nunique() # Much greater than 2.0x
20.0
>>> manycolors.memory_usage(index=False, deep=True)
6500
>>> manycolors.astype('category').memory_usage(index=False, deep=True)
585
Бонусом является и повышение эффективности вычислений: для категориальных Series строковые операции выполняются над .cat.categories атрибутом , а не над каждым исходным элементом Series.
Другими словами, операция выполняется один раз для каждой уникальной категории, а результаты отображаются обратно на значения. Категориальные данные имеют аксессуар .cat, который является окном в атрибуты и методы для манипулирования категориями:
>>> ccolors = colors.astype('category')
>>> ccolors.cat.categories
Index(['burnt orange', 'mint green', 'navy', 'periwinkle', 'rose'], dtype='object')
На самом деле, вы можете воспроизвести нечто похожее на пример выше, который вы сделали вручную:
>>> ccolors.cat.codes
0 3
1 1
2 0
3 3
4 0
5 4
6 4
7 1
8 4
9 2
dtype: int8
Все, что вам нужно сделать, чтобы в точности повторить предыдущий ручной вывод, это изменить порядок кодов:
>>> ccolors.cat.reorder_categories(mapper).cat.codes
0 0
1 1
2 2
3 0
4 2
5 3
6 3
7 1
8 3
9 4
dtype: int8
Обратите внимание, что dtype - это тип NumPy int8, 8-битное знаковое целое число, которое может принимать значения от -127 до 128. (Для представления значения в памяти требуется всего один байт. 64-битное знаковое ints было бы излишним с точки зрения использования памяти.) Наш грубый пример привел к данным int64 по умолчанию, тогда как pandas достаточно умна, чтобы привести категориальные данные к минимально возможному числовому dtype.
Большинство атрибутов для .cat связаны с просмотром и манипулированием самими базовыми категориями:
>>> [i for i in dir(ccolors.cat) if not i.startswith('_')]
['add_categories',
'as_ordered',
'as_unordered',
'categories',
'codes',
'ordered',
'remove_categories',
'remove_unused_categories',
'rename_categories',
'reorder_categories',
'set_categories']
Правда, есть несколько оговорок. Категориальные данные обычно менее гибкие. Например, при вставке ранее не встречавшихся значений необходимо сначала добавить это значение в контейнер .categories:
>>> ccolors.iloc[5] = 'a new color'
# ...
ValueError: Cannot setitem on a Categorical with a new category,
set the categories first
>>> ccolors = ccolors.cat.add_categories(['a new color'])
>>> ccolors.iloc[5] = 'a new color' # No more ValueError
Если вы планируете устанавливать значения или изменять данные, а не производить новые вычисления, Categorical типы могут оказаться менее проворными.
6. Introspect Groupby Objects via Iteration
Когда вы вызываете df.groupby('x'), получаемые панды groupby объектов могут быть немного непрозрачными. Этот объект лениво инстанцируется и сам по себе не имеет никакого осмысленного представления.
Вы можете продемонстрировать это на наборе данных abalone из примера 1:
>>> abalone['ring_quartile'] = pd.qcut(abalone.rings, q=4, labels=range(1, 5))
>>> grouped = abalone.groupby('ring_quartile')
>>> grouped
<pandas.core.groupby.groupby.DataFrameGroupBy object at 0x11c1169b0>
Хорошо, теперь у вас есть groupby объект, но что это за штука, и как ее увидеть?
Прежде чем вызывать что-то вроде grouped.apply(func), вы можете воспользоваться тем, что groupby объекты итерируемы:
>>> help(grouped.__iter__)
Groupby iterator
Returns
-------
Generator yielding sequence of (name, subsetted object)
for each group
Каждая "вещь", получаемая grouped.__iter__(), представляет собой кортеж (name, subsetted object), где name - значение столбца, по которому производится группировка, а subsetted object - DataFrame, который является подмножеством исходного DataFrame на основе любого указанного вами условия группировки. То есть данные группируются по группам:
>>> for idx, frame in grouped:
... print(f'Ring quartile: {idx}')
... print('-' * 16)
... print(frame.nlargest(3, 'weight'), end='\n\n')
...
Ring quartile: 1
----------------
sex length diam height weight rings ring_quartile
2619 M 0.690 0.540 0.185 1.7100 8 1
1044 M 0.690 0.525 0.175 1.7005 8 1
1026 M 0.645 0.520 0.175 1.5610 8 1
Ring quartile: 2
----------------
sex length diam height weight rings ring_quartile
2811 M 0.725 0.57 0.190 2.3305 9 2
1426 F 0.745 0.57 0.215 2.2500 9 2
1821 F 0.720 0.55 0.195 2.0730 9 2
Ring quartile: 3
----------------
sex length diam height weight rings ring_quartile
1209 F 0.780 0.63 0.215 2.657 11 3
1051 F 0.735 0.60 0.220 2.555 11 3
3715 M 0.780 0.60 0.210 2.548 11 3
Ring quartile: 4
----------------
sex length diam height weight rings ring_quartile
891 M 0.730 0.595 0.23 2.8255 17 4
1763 M 0.775 0.630 0.25 2.7795 12 4
165 M 0.725 0.570 0.19 2.5500 14 4
Соответственно, объект groupby также имеет .groups и группу-геттер, .get_group():
>>> grouped.groups.keys()
dict_keys([1, 2, 3, 4])
>>> grouped.get_group(2).head()
sex length diam height weight rings ring_quartile
2 F 0.530 0.420 0.135 0.6770 9 2
8 M 0.475 0.370 0.125 0.5095 9 2
19 M 0.450 0.320 0.100 0.3810 9 2
23 F 0.550 0.415 0.135 0.7635 9 2
39 M 0.355 0.290 0.090 0.3275 9 2
Это поможет вам быть немного более уверенным в том, что операция, которую вы выполняете, является именно той, которую вы хотите:
>>> grouped['height', 'weight'].agg(['mean', 'median'])
height weight
mean median mean median
ring_quartile
1 0.1066 0.105 0.4324 0.3685
2 0.1427 0.145 0.8520 0.8440
3 0.1572 0.155 1.0669 1.0645
4 0.1648 0.165 1.1149 1.0655
Независимо от того, какие вычисления вы выполняете в grouped, будь то отдельный метод pandas или пользовательская функция, каждый из этих "подкадров" передается один за другим в качестве аргумента в этот вызываемый объект. Отсюда и происходит термин "split-apply-combine": разбиваем данные на группы, выполняем вычисления для каждой группы и объединяем в некий агрегированный способ.
Если вам трудно представить, как именно будут выглядеть группы, просто пройдитесь по ним и распечатайте несколько, это может быть очень полезно.
7. Используйте этот трюк с отображением для бинирования членов
Допустим, у вас есть серия и соответствующая "таблица отображения", в которой каждое значение принадлежит к многочленной группе или вообще не принадлежит к группам:
>>> countries = pd.Series([
... 'United States',
... 'Canada',
... 'Mexico',
... 'Belgium',
... 'United Kingdom',
... 'Thailand'
... ])
...
>>> groups = {
... 'North America': ('United States', 'Canada', 'Mexico', 'Greenland'),
... 'Europe': ('France', 'Germany', 'United Kingdom', 'Belgium')
... }
Другими словами, вам нужно отобразить countries в следующий результат:
0 North America
1 North America
2 North America
3 Europe
4 Europe
5 other
dtype: object
Здесь нужна функция, похожая на pd.cut() от pandas, но для бинирования на основе категориального членства. Вы можете использовать pd.Series.map(), которую вы уже видели в примере #5, чтобы имитировать это:
from typing import Any
def membership_map(s: pd.Series, groups: dict,
fillvalue: Any=-1) -> pd.Series:
# Reverse & expand the dictionary key-value pairs
groups = {x: k for k, v in groups.items() for x in v}
return s.map(groups).fillna(fillvalue)
Это должно быть значительно быстрее, чем вложенный цикл Python по groups для каждой страны в countries.
Вот тест-драйв:
>>> membership_map(countries, groups, fillvalue='other')
0 North America
1 North America
2 North America
3 Europe
4 Europe
5 other
dtype: object
Давайте разберемся, что здесь происходит. (Примечание: это отличное место, чтобы войти в область видимости функции с помощью отладчика Python, pdb, чтобы проверить, какие переменные являются локальными для функции.)
Задача состоит в том, чтобы отобразить каждую группу в groups в целое число. Однако Series.map() не распознает 'ab' - ему нужна разбитая версия, в которой каждый символ из каждой группы сопоставлен с целым числом. Именно это и делает dictionary comprehension:
>>> groups = dict(enumerate(('ab', 'cd', 'xyz')))
>>> {x: k for k, v in groups.items() for x in v}
{'a': 0, 'b': 0, 'c': 1, 'd': 1, 'x': 2, 'y': 2, 'z': 2}
Этот словарь может быть передан в s.map() для сопоставления или "перевода" его значений в соответствующие индексы групп.
8. Поймите, как pandas использует булевы операторы
Вы, возможно, знакомы с принятым в Python старшинством операторов , где and, not и or имеют более низкий приоритет, чем арифметические операторы <, <=, >, >=, != и ==. Рассмотрим два приведенных ниже утверждения, в которых < и > имеют больший приоритет, чем оператор and:
>>> # Evaluates to "False and True"
>>> 4 < 3 and 5 > 4
False
>>> # Evaluates to 4 < 5 > 4
>>> 4 < (3 and 5) > 4
True
Примечание: Это не связано конкретно с pandas, но 3 and 5 оценивается в 5 из-за оценки короткого замыкания:
"Возвращаемым значением оператора замыкания является последний оцененный аргумент". (Источник)
pandas (и NumPy, на котором построен pandas) не использует and, or или not. Вместо них используются &, | и ~, соответственно, которые являются обычными, добросовестными операторами Python bitwise operators.
Эти операторы не "придуманы" пандой. Скорее, &, | и ~ являются допустимыми встроенными операторами Python, которые имеют более высокий (а не низкий) приоритет, чем арифметические операторы. (pandas переопределяет методы dunder, такие как .__ror__(), которые отображаются на оператор |). Чтобы пожертвовать некоторыми деталями, вы можете считать "bitwise" "elementwise" применительно к pandas и NumPy:
>>> pd.Series([True, True, False]) & pd.Series([True, False, False])
0 True
1 False
2 False
dtype: bool
Стоит понимать эту концепцию в полном объеме. Допустим, у вас есть серия, похожая на диапазон:
>>> s = pd.Series(range(10))
Я предполагаю, что вы могли видеть это исключение, поднятое в какой-то момент:
>>> s % 2 == 0 & s > 3
ValueError: The truth value of a Series is ambiguous.
Use a.empty, a.bool(), a.item(), a.any() or a.all().
Что здесь происходит? Полезно постепенно связывать выражение круглыми скобками, указывая, как Python шаг за шагом расширяет это выражение:
s % 2 == 0 & s > 3 # Same as above, original expression
(s % 2) == 0 & s > 3 # Modulo is most tightly binding here
(s % 2) == (0 & s) > 3 # Bitwise-and is second-most-binding
(s % 2) == (0 & s) and (0 & s) > 3 # Expand the statement
((s % 2) == (0 & s)) and ((0 & s) > 3) # The `and` operator is least-binding
Выражение s % 2 == 0 & s > 3 эквивалентно (или рассматривается как) ((s % 2) == (0 & s)) and ((0 & s) > 3). Это называется расширением: x < y <= z эквивалентно x < y and y <= z.
Ладно, остановитесь на этом и давайте вернемся к языку панд. У вас есть две панды Series, которые мы назовем left и right:
>>> left = (s % 2) == (0 & s)
>>> right = (0 & s) > 3
>>> left and right # This will raise the same ValueError
Вы знаете, что высказывание вида left and right проверяет на истинность как left, так и right, как в следующем:
>>> bool(left) and bool(right)
Проблема в том, что разработчики pandas намеренно не устанавливают истинностное значение (правдивость) для всей серии. Является ли серия истинной или ложной? Кто знает? Результат неоднозначен:
>>> bool(s)
ValueError: The truth value of a Series is ambiguous.
Use a.empty, a.bool(), a.item(), a.any() or a.all().
Единственное сравнение, которое имеет смысл, - это поэлементное сравнение. Вот почему, если задействован арифметический оператор, вам понадобятся круглые скобки:
>>> (s % 2 == 0) & (s > 3)
0 False
1 False
2 False
3 False
4 True
5 False
6 True
7 False
8 True
9 False
dtype: bool
Короче говоря, если вы видите ValueError вышеприведенную ситуацию с булевым индексированием, первое, что вы должны сделать, это добавить несколько необходимых круглых скобок.
9. Загрузка данных из буфера обмена
Очень часто возникает необходимость передать данные из такого места, как Excel или Sublime Text в структуру данных pandas. В идеале вы хотите сделать это, не проходя через промежуточный этап сохранения данных в файл и последующего чтения файла в pandas.
Вы можете загрузить DataFrames из буфера данных буфера обмена вашего компьютера с помощью pd.read_clipboard(). Его аргументы в виде ключевых слов передаются в pd.read_table().
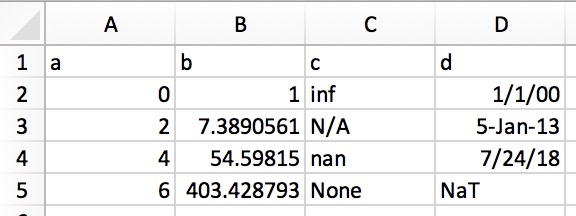
Это позволяет копировать структурированный текст непосредственно в DataFrame или Series. В Excel данные будут выглядеть примерно так:
В обычном виде (например, в текстовом редакторе) это будет выглядеть так:
a b c d
0 1 inf 1/1/00
2 7.389056099 N/A 5-Jan-13
4 54.59815003 nan 7/24/18
6 403.4287935 None NaT
Просто выделите и скопируйте обычный текст выше, и вызовите pd.read_clipboard():
>>> df = pd.read_clipboard(na_values=[None], parse_dates=['d'])
>>> df
a b c d
0 0 1.0000 inf 2000-01-01
1 2 7.3891 NaN 2013-01-05
2 4 54.5982 NaN 2018-07-24
3 6 403.4288 NaN NaT
>>> df.dtypes
a int64
b float64
c float64
d datetime64[ns]
dtype: object
10. Запись объектов pandas непосредственно в сжатый формат
Этот короткий и милый рассказ завершает список. Начиная с версии pandas 0.21.0, вы можете писать объекты pandas непосредственно для сжатия gzip, bz2, zip или xz, вместо того чтобы хранить несжатый файл в памяти и преобразовывать его. Вот пример, использующий данные abalone из трюка №1:
abalone.to_json('df.json.gz', orient='records',
lines=True, compression='gzip')
В данном случае разница в размерах составляет 11,6x:
>>> import os.path
>>> abalone.to_json('df.json', orient='records', lines=True)
>>> os.path.getsize('df.json') / os.path.getsize('df.json.gz')
11.603035760226396
Вернуться на верх